北海道大学電子科学研究所分子生命数理研究分野の李振飈准教授、小松崎民樹教授らは、一分子酵素反応の動態解析において、現在、世界中でもっともよく使われている一分子時系列解析方法は間違った解釈を与える可能性が高いことを示すことに成功しました。
酵素反応は、生体内代謝、活性調節、分子認識の仕組みを理解するうえで中心的な役割を果たすことが知られています。近年の観察技術の進歩は、反応が起っている最中に個々の酵素をリアルタイムに一分子レベルで観察することを可能にしました。近年、一分子レベルで実際に生起する酵素反応では、各素過程の反応速度定数は酵素分子がもつ多様な構造毎に異なり、酵素反応はゆっくりと変化する酵素分子の多型構造に由来して多様な時間スケールの揺らぎを持つと解釈されています(Dynamic disorderと呼ばれています)(Nature Chem. Bio. 2, 87 (2006))。
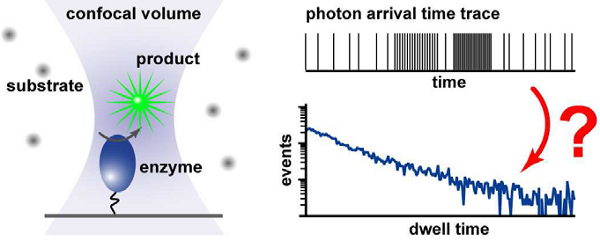
一分子蛍光分光法では、酵素反応における反応過程は、基質分子が酵素分子に結合する「オン」状態、基質分子が酵素分子から離れる「オフ」状態の2つの状態間の遷移の時系列データから評価されます(図1)。もっともよく使われている状態同定法は、時間軸に沿ったフォトン列をある一定の時間幅でビニング処理を行って蛍光強度の時系列に変換したのち、適当な閾(しきい)値を設定し、その閾値よりも高い(低い)蛍光強度をもつ時間領域をオン(オフ)状態と同定します。そして、そのオン・オフ状態の時系列データが有する(多様な時間スケールをもつ)キネティックスから酵素の構造多型性の重要性が提唱されてきました。
本研究では、計算機シミュレーションならびα-キモトリプシンの一分子観察データに対して、この従来法と(情報理論に依拠する)変化点解析を適用し、それらの結果を系統的に比較しました。その結果、シグナル/ノイズ比が無視できない場合ならびにバックグランドノイズが一定でない場合は、従来法は誤った解釈を導く可能性が極めて高いこと、dynamic disorderをもつとされていたα-キモトリプシンにおいては、その存在は実は有意に認めらないことなどを新規に明らかにしました。
本研究は、一分子動態解析に関して新しい強固な解析手法を提示すると同時に、構造多型性など一分子レベルと集団レベルとの質的な違いを正確、かつ客観的に同定できるものとして期待されています。
本研究は、カトリック大学ルーベン校Johan Hofkens教授ら(ベルギー)、ラドボード大学・分子物質研究所 Kerstin Blank 教授ら(オランダ)との共同研究で行われました。本研究成果は,米国科学誌『ACS Nano』で2012年1月24日(日本時間)に公開され、1月号のハイライトに取り上げられました。