北海道大学電子科学研究所(三澤弘明所長)分子生命数理研究分野の李振飈准教授、小松崎民樹教授らの研究グループは、(例えば、開いているか/閉じているか、結合しているか/離れているか、といった)有限個で連続的でない飛び飛びの値をもつ分子の時系列データから、分子の状態をネットワークという表現を用いて客観的に評価し、分子が将来取り得るデータ出力を予想する新しい解析手法を開発することに成功!
通常、医師は患者の心拍数の時系列データを測ることにより患者の病状を診察したりする。仮に、その時系列データ以外の、例えば、性別、年齢、体温、既往症などの如何なる情報も得られないとしたとき、どのようにして客観的に患者の状態を判断できるのであろうか?
李准教授らは、分子の状態を(WWWや人の繋がりのネットワークなどと同じ)ネットワークという表現を用いて表し、分子の時系列データから、ネットワークを構成するノード(WWWではURL、人間社会では人)がどれくらいの数存在し、どのノードとどのノードが強くつながっているかを客観的に評価する数学的な手法を開発した。時系列データは一次元の、非常に限定された情報であるため、それらの評価には恣意性が入り込む余地が高いことが知られている。李准教授らは、考え得るすべてのネットワークにおいて、観測されたデータが保証していない情報がどれくらい組み込まれているかを定量化する新しい指標を考案し、その指標を最小化する(データに忠実、かつ最も客観的な)ネットワークを時系列データから抽出するアルゴリズムを開発することに成功した。李准教授らは、この手法を一分子酵素反応の一分子計測データ(Englishら Nature Chem. Bio. 2006)の解析に適用した。その結果、反応する相手の基質分子の濃度に依存してネットワークが動的に変化することや、最小限の過剰情報量をもつネットワークを導入することによって、どのようなデータを取得すると正しいネットワークに最も近い答えを導き得るかに関する知見が得られることなどを明らかにした。
現在、この解析手法は分子の時系列データから、分子の状態を、できるだけ主観を排除して、同定し、分子が将来取り得る状態を予想するとともに、実際にデータを計測する実験家に対してデータ取得上の新たな指針の提案にも繋がるものとして期待されている。
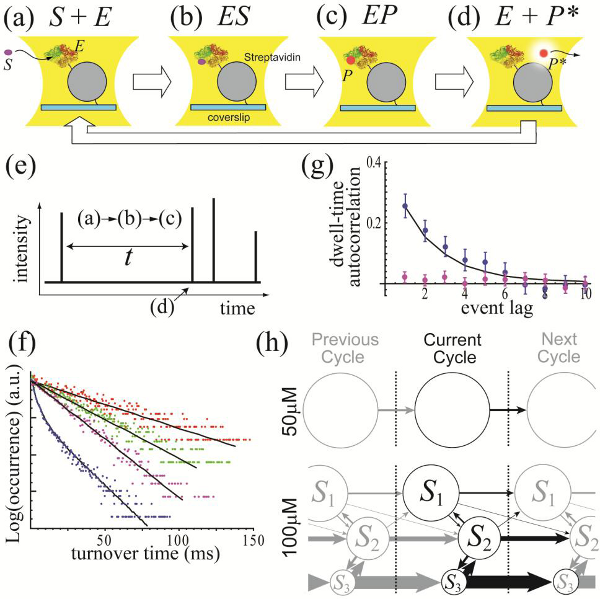
詳細な説明:近年の一分子実験の進展により、酵素触媒反応における酵素-基質相互作用の一分子レベルでの実時間追跡が可能となってきた。ミカエリス・メンテン反応におけるDynamic disorderとは一分子レベルにおいて非指数型キネティックスが顕在化することを意味する。非指数型キネティックスは酵素の構造間遷移の時間スケールとE+S⇔ES→E+Pの反応時間スケールが拮抗する濃度領域で生じる。その濃度領域では、酵素が完全に構造緩和する前に次の反応が生起するため、構造の記憶が反応のキネティックスに影響を及ぼすためである(e.g., Nat. Chem. Bio., 2, 87 (2006))。このDynamic disorderの起源を論じるうえで、観測された時系列そのものから背後に存在する状態遷移ネットワーク、キネティックスキーム(KS)、が抽出できれば、極めて有用な情報を与えるものと期待される。しかしながら、一般に、図1 (e)に示すような、dwell-time 時系列データが与えられたとき、自己相関関数などのキネティックスを再現するキネティックスキーム(KS)は原理的に無数に存在する。換言すると、dwell-time 時系列データの統計的な相関情報から KS を一義的に同定することができない。我々は「すべての可能なKSのなかで、同等の最大予測性能をもち、最小かつ客観的 (unbiased) な KS をデータから抽出する方法論を新規に開発した。
図1にEscherichia coli β-galactosidaseの一分子酵素ターンオーバー実験により得られたdwell-time 時系列データに対する我々の方法論を適用した結果を示す。(a)-(d)に一分子酵素反応実験の概略を、(e)には蛍光発光する生成物が共焦点領域に存在している間だけ光子が観測されるバースト時系列を示している。バースト間の “dark” dwell time t は触媒反応のターンオーバー時間に対応している。(f)は異なる基質濃度[S]におけるターンオーバー時間のLog-linearヒストグラムを示しており、基質濃度100μMでは非指数型キネティックスに移行することが分かる。(g)は50μMおよび100μMにおけるDwell timeの自己相関関数を示しており、100μMでは、顕著な分子記憶が存在することが分かる。(h)はデータ駆動的に抽出された 50μM および 100μM におけるKSを示している。現在のターンオーバーサイクルの「状態」(円)およびその状態からの遷移(矢印)を黒の実線で、前回および次回のサイクルのものを灰色の実線で表している。円の面積は相対的な滞在確率の大きさ、矢印の太さは遷移確率の大きさを反映している。また、ここでは、簡単のために、各状態からの遷移のうち、自分自身への状態遷移は省略している。(f)および(g)における実線はこれらのKSから求められた解析解を意味しており、基質濃度依存の実験データを忠実に再現していることが分かる。データ駆動的に再構成されたKSは、様々な基質濃度に対する一分子酵素反応の分子記憶、非指数型キネティックス(dynamic disorder)の起源を論じるうえで、背後に存在する状態と状態間の遷移の繋がりに関する重要な情報を提供するものである。
本研究成果は文部科学省科研費・新学術領域「少数性生物学」、ヒューマン・フロンティア・サイエンス・プログラム(本部:フランス・ストラスブール)の支援を受けました。本研究成果は『Phys. Rev. Lett.』誌に 8月 2日付(電子版 8月 1日付)で公開されました。 “Aggregated Markov Model Using Time Series of Single Molecule Dwell Times with Minimum Excessive Information,” C.B. Li and T. Komatsuzaki, Phys. Rev. Lett., in press, 2013.